2001년 9월 1일 – 2006년 2월 28일 : University of Wisconsin-Madison, Dept. of Biochemistry, Ph.D.
1999년 3월 2일 – 2001년 2월 26일 : 서울대학교 자연과학대학 화학과, 이학석사(생화학전공)
1992년 3월 2일 – 1999년 2월 26일 : 서울대학교 사범대학 화학교육과, 이학사
2020년 3월 2일 – 현재 : 서울대학교 약학대학 약학과, 교수
2022년 8월 20일 – 2024년 8월 19일 : 서울대학교 호암교수회관 집행이사(관장)
2021년 7월 27일 – 2022년 8월 19일 : 서울대학교 약학대학 학생부학장
2016년 1월 10일 – 2017년 1월 9일 : The Scripps Research Institute (TSRI), Visiting Scientific Collaborator
2013년 9월 1일 – 2020년 2월 28이 : 서울대학교 약학대학 약학과, 부교수
2009년 8월 1일 – 2013년 8월 31일 : 서울대학교 약학대학 약학과, 조교수
2006년 3월 1일 – 2009년 2월 28일 : The Scripps Research Institute (TSRI), Research Associate
2001년 3월 2일 – 2001년 7월 31일 : 서울대학교 자연과학대학 화학과, 연구원(석사급)
- Structure-function relationships of biomedically important proteins
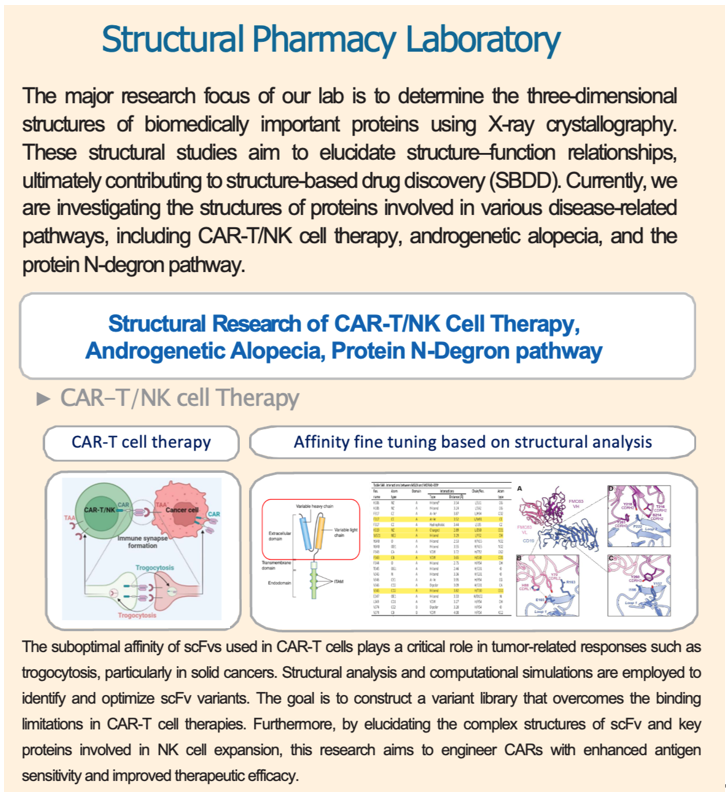
- Structural research of CAR-T/NK cell therapy
- Structural research of androgenetic alopecia
- Structural research of the N-degron pathway in protein degradation
- 바이오데이터 전문연구인력 양성
-
1. “Structural insight into the distinct regulatory mechanism of the HEPN–MNT toxin–antitoxin system in Legionella pneumophila” Chenglong Jin*, Cha-Hee Jeon*, Heung Wan Kim, Jin Mo Kang, Yuri Choi, Sung-Min Kang, Hyung Ho Lee, Do-Hee Kim†, Han,B.W.†, Bong-Jin Lee†(2024) Nature Communications. 2024 Nov 24;15(1):10188. doi: 10.1038/s41467-024-54551-0. PMID: 39582057(* equally contributed, † co-corresponding authors)
2. “Structural study for substrate recognition of human N-terminal glutamine amidohydrolase 1 in the arginine N-degron pathway” Kang JM, Park JS, Lee JS, Jang JY, Han,B.W.(2024)Protein Science. 2024 Jul;33(7):e5067. doi: 10.1002/pro.5067. PMID: 38864716
3. “Structural insights into apoptotic regulation of human Bfk as a novel Bcl-2 family member” Jang DM*, Oh EK*, Hahn H, Kim HS†, and Han,B.W.† (2022) Computational and Structural Biotechnology Journal. 2022 Jan 28;20:745-756. doi: 10.1016/j.csbj.2022.01.023. eCollection 2022. PMID: 35140891 (* equally contributed, † co-corresponding authors)
4. “Structural basis for SdgB- and SdgA-mediated glycosylation of staphylococcal adhesive proteins” Kim DG*, Baek I*, Lee Y, Kim H, Kim JY, Bang G, Kim S, Yoon HJ, Han,B.W.†,Suh SW†, and Kim HS†. (2021) Acta Crystallographica Section D Structural Biology. 2021 Nov 1;77(Pt 11):1460-1474. doi: 10.1107/S2059798321010068. Epub 2021 Oct 20.PMID: 34726173 (* equally contributed, † co-corresponding authors)
5. “Differential Effects of Cancer-Associated Mutations Enriched in Helix H3 of PPARγ” Jang DM*, Jang JY*, Kim HJ, and Han,B.W.(2020) Cancers (Basel). 2020 Nov 30;12(12):3580. doi: 10.3390/cancers12123580. PMID: 33266062 (* equally contributed)
6. “Structural Insight on Functional Regulation of Human MINERVA Protein” Hahn H, Lee DE, Jang DM, Kim J, Lee Y, Cheong H, Han,B.W.†, and Kim HS†. (2020) International Journal of Molecular Sciences. 2020 Oct 31;21(21):8186. doi: 10.3390/ijms21218186. PMID: 33142954 († co-corresponding authors)
7. “Novel HDAC Inhibitor MAKV-8 and Imatinib Synergistically Kill Chronic Myeloid Leukemia Cells via Inhibition of BCR-ABL/MYC-signaling: Effect on Imatinib Resistance and Stem Cells” Lernoux M, Schnekenburger M, Losson H, Vermeulen K, Hahn H, Gérard D, Lee JY, Mazumder A, Ahamed M, Christov C, Kim DW, Dicato M, Bormans G, Han,B.W.†, and Diederich M†. (2020) Clinical Epigenetics. 2020 May 19;12(1):69. doi: 10.1186/s13148-020-00839-z. PMID: 32430012 († co-corresponding authors)
8. “Cyclin-Dependent Kinase 5 Inhibitor Butyrolactone I Elicits a Partial Agonist Activity of Peroxisome Proliferator-Activated Receptor γ” Ahn S*, Jang DM*, Park SC, An S, Shin J, Han,B.W.†, and Noh M.† (2020) Biomolecules. 2020 Feb 11;10(2). pii: E275. doi: 10.3390/biom10020275. PMID: 32054125 (* equally contributed, † co-corresponding authors)
9. “Structural Analyses on the Deamidation of N-Terminal Asn in the Human N-Degron Pathway” Park JS, Lee JY, Nguyen YTK, Kang NW, Oh EK, Jang DM, Kim HJ, Kim DD, and Han,B.W.(2020) Biomolecules. 2020 Jan 20;10(1). pii: E163. doi: 10.3390/biom10010163. PMID: 31968674
10. “Structural and Biophysical Analyses of Human N-Myc Downstream-Regulated Gene 3 (NDRG3) Protein” Kim KR, Kim KA, Park JS, Jang JY, Choi Y, Lee HH, Lee DC, Park KC, Yeom YI, Kim HJ, and Han,B.W.(2020) Biomolecules. 2020 Jan 6;10(1). pii: E90. doi: 10.3390/biom10010090. PMID: 31935861
11. “Structural and Functional Analyses of Human ChaC2 in Glutathione Metabolism” Nguyen YTK, Park JS, Jang JY, Kim KR, Vo TTL, Kim KW, and Han,B.W.(2019) Biomolecules. 2019 Dec 24;10(1). pii: E31. doi: 10.3390/biom10010031. PMID: 31878259
12. “The DRS–AIMP2–EPRS subcomplex acts as a pivot in the multi-tRNA synthetase complex” Hahn H*, Park SH*, Kim HJ, Kim S, and Han,B.W.(2019) IUCr Journal. 2019 Aug 24;6(Pt 5):958-967. doi: 10.1107/S2052252519010790. eCollection 2019 Sep 1., PMID: 31576228 (* equally contributed)
13. “Structural basis for the inhibitory effects of a novel reversible covalent ligand on PPARγ phosphorylation” Jun Young Jang*, Hyun Soo Kim*, Hyun-Jung Kim, Se Won Suh, Seung Bum Park†, and Han,B.W.†, (2019) Scientific Reports, 2019 Aug 1;9(1):11168. doi: 10.1038/s41598-019-47672-w. PMID: 31371757 (* equally contributed, † co-corresponding authors)
14. “Structure and immunogenicity of a stabilized HIV-1 envelope trimer based on a group-M consensus sequence” Sliepen K*, Han,B.W.†, Bontjer I*, Mooij P, Garces F, Behrens AJ, Rantalainen K, Kumar S, Sarkar A, Brouwer PJM, Hua Y, Tolazzi M, Schermer E, Torres JL, Ozorowski G, van der Woude P, de la Peña AT, van Breemen MJ, Camacho-Sánchez JM, Burger JA, Medina-Ramírez M, González N, Alcami J, LaBranche C, Scarlatti G, van Gils MJ, Crispin M, Montefiori DC, Ward AB, Koopman G, Moore JP, Shattock RJ, Bogers WM, Wilson IA†, and Sanders RW†. (2019) Nature Communications, 2019 May 29;10(1):2355. doi: 10.1038/s41467-019-10262-5. PMID: 31142746 (* equally contributed, † co-corresponding authors)
15. “Unique N-terminal extension domain of human asparaginyl-tRNA synthetase elicits CCR3-mediated chemokine activity” Park JS, Park MC, Lee KY, Goughnour PC, Jeong SJ, Kim HS, Kim HJ, Lee BJ, Kim S, and HanB.W.(2018) International Journal of Biological Macromolecules, 2018 Dec;120(Pt A):835-845. doi: 10.1016/j.ijbiomac.2018.08.171. Epub 2018 Aug 30. PMID: 30171954
16. “Structural basis for the substrate recognition of peptidoglycan pentapeptides by Enterococcus faecalis VanYB” Kim HS*†, Hahn H*, Kim J, Jang DM, Lee JY, Back JM, Im HN, Kim H, Han,B.W.†, and Suh SW†. (2018) International Journal of Biological Macromolecules, 2018 Nov;119:335-344. doi: 10.10
-
17. “Antigen recognition by variable lymphocyte receptors” Han, B.W., Herrin, B.R., Cooper, M.D. & Wilson, I.A., (2008) Science, 321(5897):1834-7, 2008.09.26.
18. “Membrane association, mechanism of action, and structure of Arabidopsis EMBRYONIC FACTOR 1 (FAC1)” Han, B.W., Bingman, C.A., Mahnke, D.K., Bannen, R.M., Sabina, R.L. & Phillips, G.N., Jr. (2006) Journal of Biological Chemistry, 281(21):14939-47, 2006. 05. 26.
강진모 (박사, 2025년 2월 졸업)
장동만 (박사, 2021년 2월 졸업)
김경록 (박사, 2020년 2월 졸업)
한형구 (박사, 2020년 2월 졸업)
Nguyen Thi Kim Yen (박사, 2020년 2월 졸업)
박준성 (박사, 2019년 2월 졸업)
박상호 (박사, 2016년 2월 졸업)
문성희 (석사, 2025년 2월 졸업)
이슬희 (석사, 2024년 8월 졸업)
서주희 (석사, 2023년 2월 졸업)
임혜원 (석사, 2023년 2월 졸업)
황은미 (석사, 2022년 8월 졸업)
이재석 (석사, 2020년 8월 졸업)
오은경 (석사, 2020년 2월 졸업)
박준성 (박사, 2019년 2월 졸업)
오서영 (석사, 2019년 2월 졸업)
김민주 (석사, 2017년 8월 졸업)
박상호 (박사, 2016년 2월 졸업)
백인화 (석사, 2014년 8월 졸업)
김경록 (석사, 2014년 2월 졸업)
백장미 (석사, 2013년 2월 졸업)
이지연 (석사, 2013년 2월 졸업)
박미설 (석사, 2012년 2월 졸업)
한미라 (석사, 2012년 2월 졸업)

02-762-8322


